英國變種新冠病毒(正式名稱:VOC 202012/01)Q&A
新冠肺炎病毒(SARS-CoV-2)對全球社會帶來巨變。本院根據全球共享流感數據倡議組織(GISAID)資料,分析二十萬筆以上的病毒基因序列樣本資料。發現病毒可歸類為六大類型。其中「第六型」自今(2020)年2、3月開始在各國急速竄升,4月已雄霸一方,佔目前流傳病毒類型的99%。
然而,病毒變異仍是現在進行式,尤其近日引發討論的英國變種病毒現在狀況如何?又會如何發展?本院除試著回答這些問題外,也將持續透過此監測網觀察新冠肺炎病毒的變異走向。
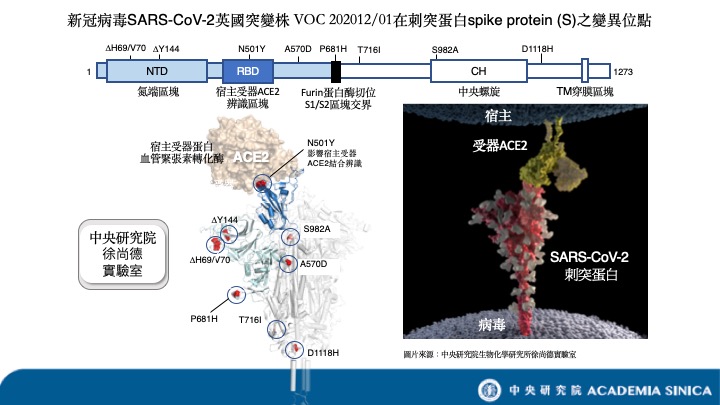
VOC 202012/01涉及的變異位點眾多,後續各單位在認定病毒株是否為VOC 202012/01時,標準不盡相同(註一)。以VOC 202012/01典型病毒株EPI_ISL_601443作為比對標準,根據全球共享流感數據倡議組織(GISAID)的265,138株病毒株基因體資料,可以發現VOC 202012/01有1,354株來自英國,有3株來自丹麥,這兩國發現第一株的時間分別是9月20日、11月9日。發生率在英國自2020年10月底開始攀升。
表一所列的17個變異包括(以下變異名稱的描述先呈現核苷酸變異,括號內再補充氨基酸變異名稱):
- ORF1ab蛋白上3個單位點(SNV)變異:C3267T(T1001I)、C5388A(A1708D)、T6954C(I2230T)。
- ORF1ab蛋白上1個缺失(deletion)變異:核苷酸11288 – 11296 deletion(SGF 3675 – 3677 deletion)。
- S蛋白上2個缺失變異:核苷酸21765 – 21770 deletion(HV 69 – 70 deletion)、核苷酸21991 – 21993 deletion(Y144 deletion)。
- S蛋白上6個單位點變異:A23063T(N501Y)、C23271A(A570D)、C23604A(P681H)、C23709T(T716I)、T24506G(S982A)、G24914C(D1118H)。
- Orf8蛋白上3個單位點變異:C27972T(Q27stop)、G28048T(R52I)、A28111G(Y73C)。
- N蛋白上2個核苷酸變異:28280 GAT->CTA(D3L)、C28977T(S235F)。
- ORF1ab上5個單位點變異:C913T、C5986T、C14676T、C15279T、C16176T。
- M基因上1個單位點變異:T26801C。
我們根據GISAID的265,138株病毒株基因體資料來比對,若是單以S蛋白上6個單位點變異作為標準,可以發現VOC 202012/01有1,460株來自英國,3株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、11月9日、11月30日。若是單以S蛋白上2個缺失變異作為標準,可以發現VOC 202012/01有1,463株來自英國,13株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、10月12日、11月30日,另外還有一株來自斯洛維尼亞,時間是3月07日(目前無法排除是否GISAID資料的問題,但若資料正確,那此變異很早就發生過)。若是單以S蛋白上2個缺失(deletion)變異加上6個單位點(SNV)變異作為標準,可以發現VOC 202012/01有1,445株來自英國,3株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、11月9日、11月30日。若是把文件表一所列的17個變異都納入,可以發現VOC 202012/01有1,354株來自英國,3株來自丹麥,第一株的時間分別是9月20日、11月9日。最後,若是進一步把23個病毒基因變異點全部納入,只發現EPI_ISL_601443(9月20日)最接近,完整攜帶文件表一所列的17個變異,但6個同義單位點變異上只攜帶5個變異,在核苷酸16176位點上是攜帶C而非變異T。無法找到滿足全部23個變異的可能解釋,因為VOC 202012/01屬於GISAID上的B1.1.7支系,EPI_ISL_601443(9月20日)為該支系典型病毒株,缺少的變異會在同屬B1.1.7支系的其他病毒株中發現。
表一所列的17個變異包括(以下變異名稱的描述先呈現核苷酸變異,括號內再補充氨基酸變異名稱):
- ORF1ab蛋白上3個單位點(SNV)變異:C3267T(T1001I)、C5388A(A1708D)、T6954C(I2230T)。
- ORF1ab蛋白上1個缺失(deletion)變異:核苷酸11288 – 11296 deletion(SGF 3675 – 3677 deletion)。
- S蛋白上2個缺失變異:核苷酸21765 – 21770 deletion(HV 69 – 70 deletion)、核苷酸21991 – 21993 deletion(Y144 deletion)。
- S蛋白上6個單位點變異:A23063T(N501Y)、C23271A(A570D)、C23604A(P681H)、C23709T(T716I)、T24506G(S982A)、G24914C(D1118H)。
- Orf8蛋白上3個單位點變異:C27972T(Q27stop)、G28048T(R52I)、A28111G(Y73C)。
- N蛋白上2個核苷酸變異:28280 GAT->CTA(D3L)、C28977T(S235F)。
- ORF1ab上5個單位點變異:C913T、C5986T、C14676T、C15279T、C16176T。
- M基因上1個單位點變異:T26801C。
我們根據GISAID的265,138株病毒株基因體資料來比對,若是單以S蛋白上6個單位點變異作為標準,可以發現VOC 202012/01有1,460株來自英國,3株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、11月9日、11月30日。若是單以S蛋白上2個缺失變異作為標準,可以發現VOC 202012/01有1,463株來自英國,13株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、10月12日、11月30日,另外還有一株來自斯洛維尼亞,時間是3月07日(目前無法排除是否GISAID資料的問題,但若資料正確,那此變異很早就發生過)。若是單以S蛋白上2個缺失(deletion)變異加上6個單位點(SNV)變異作為標準,可以發現VOC 202012/01有1,445株來自英國,3株來自丹麥,1株來自澳洲,這三國發現第一株的時間分別是9月20日、11月9日、11月30日。若是把文件表一所列的17個變異都納入,可以發現VOC 202012/01有1,354株來自英國,3株來自丹麥,第一株的時間分別是9月20日、11月9日。最後,若是進一步把23個病毒基因變異點全部納入,只發現EPI_ISL_601443(9月20日)最接近,完整攜帶文件表一所列的17個變異,但6個同義單位點變異上只攜帶5個變異,在核苷酸16176位點上是攜帶C而非變異T。無法找到滿足全部23個變異的可能解釋,因為VOC 202012/01屬於GISAID上的B1.1.7支系,EPI_ISL_601443(9月20日)為該支系典型病毒株,缺少的變異會在同屬B1.1.7支系的其他病毒株中發現。
此外,病毒入侵細胞主要是靠病毒的刺突蛋白(Spike protein),這是病毒用來解鎖、進攻人體的鑰匙,其中刺突蛋白上的受體結合域(receptor-binding domain, RBD)是病毒最先接觸人體細胞表面的部分,若有變動,理論上便有少許可能增強病毒入侵的機率,或逃避疫苗的機會。但仍須驗證。
此次英國新變種病毒的基因共有23個變異,其中有8個(包括2個缺失(deletion)變異和6個單位點(SNV)變異)落在刺突蛋白上。6個SNV變異中又有1個在關鍵的RBD區域,也就是新冠病毒與人類細胞的結合區,是病毒進入宿主細胞的關鍵,須持續追蹤觀察。
參考資料
- Chand, Meera; Hopkins, Susan; Dabrera, Gavin; Achison, Christina; Barclay, Wendy; Ferguson, Neil; Volz, Erik; Loman, Nick; Rambaut, Andrew; Barrett, Jeff (21 December 2020). Investigation of novel SARS-COV-2 variant: Variant of Concern 202012/01 (PDF) (Report). Public Health England. Retrieved 22 December 2020. [Link]
